深圳欣博盛生物科技有限公司品牌商
16 年
手机商铺
商家活跃:
产品热度:
- NaN
- 0
- 0
- 2
- 2
全部产品
- 查看全部分类
- 欣博盛ELISA 试剂盒
- Vectorlabs
- GeneTex
- Peprotech
- Invivogen
- BioXcell
- FuGENE
- Prozyme
- Cayman
- Mirusbio
- ENZO(Alexis, Biomol, Assaydesign&stressgen)
- AbD Serotec
- Surmodics
- Millipore
- Metasystems
- Mabtech
- Proimmune
- Adipogen
- Bioporto
- Southern Biotech (SBA)
- Everest Biotech
- Chondrex
- IQ Products
- Atlas
- Detroit R&D
- StressMarq
- Cell Biolabs
- StrigoLab
- Synaptic Systems
- BioInnovatise
- Cosmo Bio
- Abwiz Bio
- Gerbu
- 蛋白
- 试剂类
- 抗体
- 分子化合物
- Cargille
- Antagen
- Nanoprobes
- Funakoshi
- Demeditec
- Enzo
- Fitzgerald
- Biovendor
- ICL
- Matreya
- PhaRNA
- Funakoshi
- Promise Advanced Proteomics
- Bioassay works
- NSJ Bio
- Aldevron
- CalBioreagents
- 3Helix
- 自产试剂盒
- TriLink
- Virostat
- Agilent
- Epicypher
- Jackson ImmunoResearch
推荐产品
公司新闻/正文
热烈祝贺ActiveMotif公司NOMe-Seq荣获《The Scientist》2013生命科学10大创新产品!
927 人阅读发布时间:2013-12-09 13:53
 ActiveMotif公司NOMe-Seq荣获《The Scientist》2013生命科学10大创新产品,NOMe-Seq是世界上首个可以分析同一DNA分子上多种表观修饰关系的试剂盒,通过高分辨率的核小体定位和甲基化谱测序,可对同一条DNA链进行核小体足迹和DNA甲基化谱分析。该方法使得研究者研究他们感兴趣的目的基因在发生DNA甲基化的情况下核小体的定位成为可能。
ActiveMotif公司NOMe-Seq荣获《The Scientist》2013生命科学10大创新产品,NOMe-Seq是世界上首个可以分析同一DNA分子上多种表观修饰关系的试剂盒,通过高分辨率的核小体定位和甲基化谱测序,可对同一条DNA链进行核小体足迹和DNA甲基化谱分析。该方法使得研究者研究他们感兴趣的目的基因在发生DNA甲基化的情况下核小体的定位成为可能。
NOMe-Seq概述
使用GpC甲基转移酶处理固定的染色质,使未被核小体以及其他结合蛋白保护的GpC二核苷发生甲基化;由于基因组中含有大量的GpC,通过酶的处理可对核小体足迹进行高分辨的定位;处理过的DNA然后经过重亚硫酸盐转化和特定基因位点测序来建立该DNA单链的甲基化谱。因为哺乳动物基因组的GpC二核苷是非甲基化的,从而内源的CpG甲基化可以同人为处理的GpC甲基化区分开来。结合GpC和CpG位点的测序结果,可确定特定基因位点上核小体定位和DNA甲基化的情况。
NOMe-Seq 优点
- 可对同一条DNA链同时进行核小体定位和CpG 甲基化分析
- 可了解DNA甲基化与核小体占位之间的瞬时关系
- 足够的灵敏度可用于分析核小体位置上微妙的变化
- 核酸酶消化分析表明无核小体占位倾向
- 染色质的固定同时提供了转录因子结合位点的信息
NOMe-Seq的工作原理
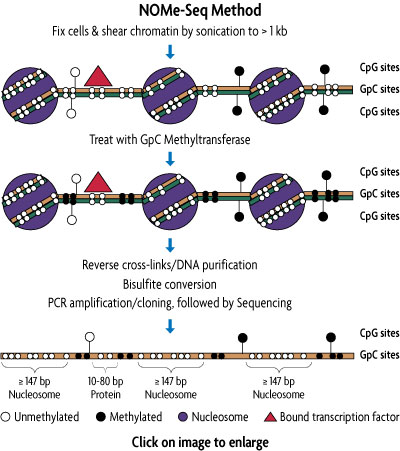
1.细胞经甲醛固定,可以保护核小体以及DNA/蛋白的相互结合。超声处理染色质产生1kb左右大小的片段;
2. 用GpC DNA甲基转移酶处理,使未被核小体以及其他结合蛋白保护的GpC二核苷发生甲基化。GpC二核苷在DNA中远远多于CpG二核苷,更适合用于核小体的定位,而且在哺乳动物细胞基因组中本身CpG是非甲基化的,因此这种人为的GpC甲基化处理并不会干扰CpG甲基化谱的建立;
3. 经过解交联和DNA纯化,用重亚硫酸盐转化GpC甲基化处理的DNA。首先DNA变性成单链,然后用重亚硫酸盐处理,未甲基化的胞嘧啶转变成尿嘧啶,而甲基化的胞嘧啶不发生变化,经PCR扩增、克隆和测序进行基因特异性分析;
4. 对测序结果作图来分析比较人为的甲基化GpC和内源的甲基化CpG。含未甲基化GpC的约147bp的区域表明为核小体所在位置,约10-80bp的含未甲基化GpC的区域显示为蛋白结合位点。对GpC甲基化和CpG甲基化测序结果重叠分析,可了解DNA甲基化与核小体占位之间的瞬时关系。阴性对照可帮助解决内源或人为的GpCpG三核苷甲基化带来的影响。已验证NOMe-Seq可通过传统的Sanger测序法或Illumina®第二代基因测序法来进行基因特异性分析。
NOMe-Seq产品信息
NOMe-Seq试剂盒提供足够的试剂可用于10次GpC甲基转移酶处理和10次阴性对照反应。这些试剂可用于染色质固定、核蛋白提取、GpC甲基转移酶处理、染色质解交联、重亚硫酸盐转化和DNA纯化。试剂盒提供处理250,000个细胞的优化操作步骤以及PCR扩增、DNA克隆和测序的操作指南。
| 产品名称 | 规格 | 产品编号 |
|---|---|---|
| NOMe-Seq | 10 rxns | 54000 |
NOMe-Seq部分引用文献
“H2A.Z Maintenance dufing Mitosis Reveals Nucleosome Shifting on Mitotically Silenced Genes.” by Kelly, T.K. et al. (2010) Mol. Cell, 39(6): 901-911.
“OCT4 establishes and maintains nucleosome-depleted regions that provide additional layers of epigenetic regulation of its target genes.” by You, J.S. et al. (2011) Proc. Natl. Acad. Sci., 108(35): 14497-14502.
“Dynamic Nucleosome-Depleted Regions at Androgen Receptor Enhancers in the Absence of Ligand in Prostate Cancer Cells.” by Andreu-Vieyra, C. et al. (2011) Mol. Cell Biol., 31(23): 4648-4662.
“Genome-wide mapping of nucleosome positioning and DNA methylation within individual DNA molecules.” by Kelly, T.K. et al. (2012) Genome Res., doi:10.1101/gr.143008.112.
“Hypomethylation of a LINE-1 Promoter Activates an Alternate Transcript of the MET Oncogene in Bladders with Cancer.” by Wolff, E. M. et al. (2010) PLoS Genetics, 6(4): e1000917.
“DNA methylation directly silences genes with non-CpG island promoters and establishes a nucleosome occupied promoter” by Han Han et al. (2011) Hum. Mol. Genet., 20(22): 4299-4310.
“Polycomb Repressed Genes have Permissive Enhancers that Initiate Reprogramming.” by Taberlay, P.C. et al. (2011) Cell, 147(6): 1283-1294.
更多产品信息请联系ActiveMotif中国战略合作伙伴——欣博盛生物,全国服务热线4006-800-892。






